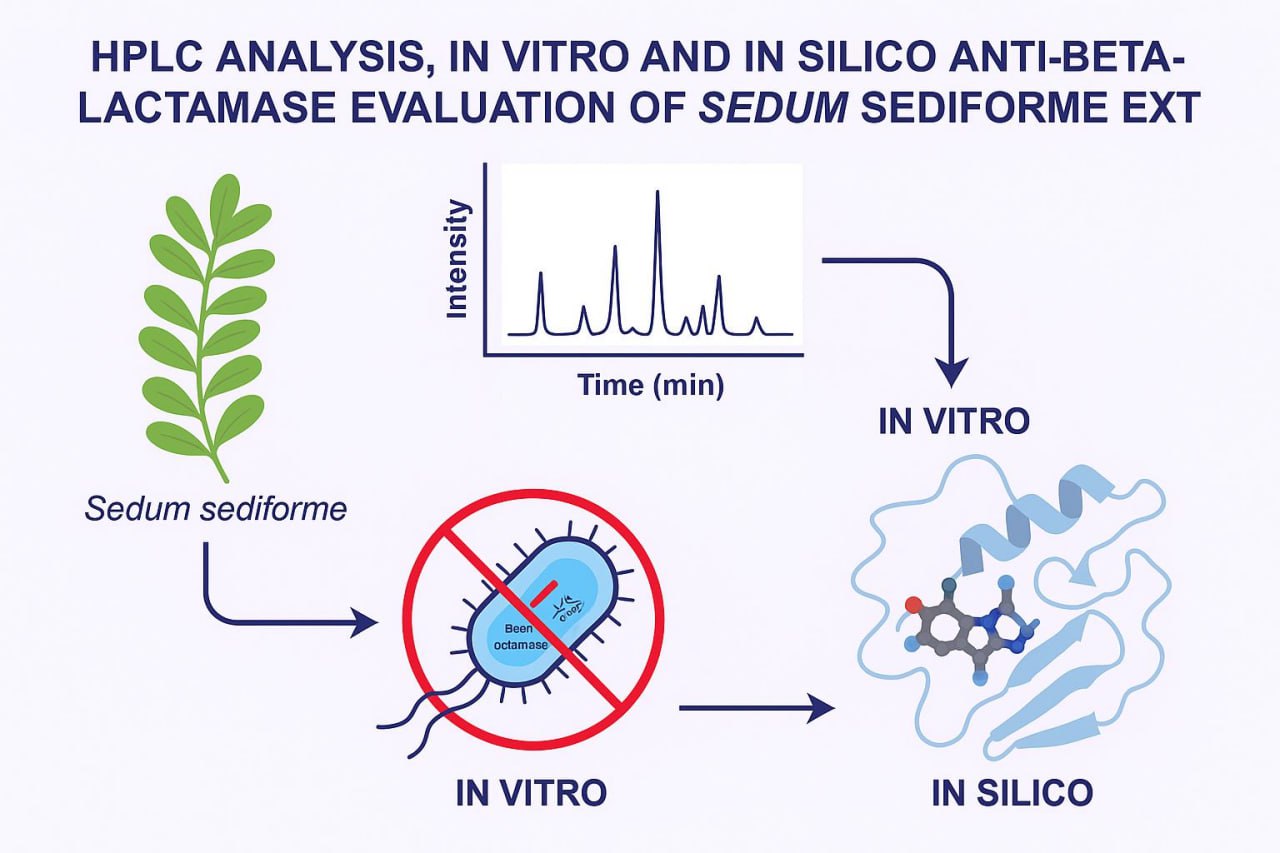
Phytochemical Characterization and Evaluation of Sedum sediforme Extracts as Potential β-lactamase Inhibitors: An In vitro and In Silico Study
DOI:
https://doi.org/10.29356/jmcs.v70i1.2525Keywords:
β-lactamases, Sedum sediforme, molecular docking, HPLC, Swiss ADMEAbstract
Abstract. This study investigated the flavonoid content and β-lactamase inhibitory activity of three Sedum sediforme extracts: crude (CrE), chloroform (ChE), and ethyl acetate (EAe). Total flavonoids were quantified using AlCl3 complexation, and HPLC analysis revealed quercetin (36.52 %) and gallic acid (24.11 %) as the predominant compounds in CrE. Enzymatic assays showed that CrE exhibited the highest β-lactamase inhibition, followed by ChE and EAe. In addition, an in silico analysis was conducted to explore the molecular interactions between phenolic compounds from S. sediforme and various β-lactamase enzymes. Seventeen phenolic constituents were identified by HPLC, with notable levels of caffeic acid (6.65 %), hesperetin (6.17 %), syringic acid (5.47 %), kaempferol (4.05 %), and rutin (3.83 %). Three-dimensional structures of these compounds were obtained from PubChem, optimized using Avogadro, and docked against four β-lactamase targets—TEM-1 (PDB: 1NYM), NDM-1 (PDB: 4EXS), AmpC (PDB: 1C3B), and OXA-48 (PDB: 7KHQ)—via AMDock. Docking results revealed strong binding affinities, including quercetin with TEM-1 (–8.9 kcal/mol), rutin with AmpC (–9.3 kcal/mol) and NDM-1 (–6.79 kcal/mol), and gallic acid with OXA-48 (–7.45 kcal/mol). Interaction profiling using BIOVIA Discovery Studio confirmed hydrogen bonding, hydrophobic interactions, and steric complementarity. A significant correlation was found between compound concentration and binding energy for TEM-1 (p = 0.023) and AmpC (p = 0.010). Pharmacokinetic predictions from Swiss ADME showed that quercetin and gallic acid satisfy Lipinski’s Rule of Five, indicating good oral bioavailability, whereas rutin does not. BOILED-Egg analysis predicted blood–brain barrier permeability for quercetin and gallic acid. Toxicity predictions using ProTox-II revealed potential organ-specific toxicities among top ligands.
Resumen. En este estudio se investigó el contenido de flavonoides y la actividad inhibidora en β-lactamasas de tres extractos de Sedum sediforme: crudo (CrE), cloroformo (ChE) y acetato de etilo (EAe). Los flavonoides totales se cuantificaron mediante la formación de complejos con AlCl₃, y su análisis mediante HPLC reveló que la quercetina (36.52 %) y el ácido gálico (24.11 %) fueron los compuestos predominantes en CrE. Los ensayos enzimáticos mostraron que el extracto CrE presentó la mayor inhibición de β-lactamasas, seguida por los extractos de ChE y EAe. Además, se realizó un análisis in silico para explorar las interacciones moleculares entre los compuestos fenólicos de S. sediforme y diversas enzimas β-lactamasas. Se identificaron diecisiete componentes fenólicos mediante HPLC, con concentraciones notables de ácido cafeico (6.65 %), hesperetina (6.17 %), ácido siríngico (5.47 %), kaempferol (4.05 %) y rutina (3.83 %). Las estructuras tridimensionales de estos compuestos se obtuvieron de PubChem, se optimizaron con Avogadro y se acoplaron a cuatro blancos de β-lactamasa: TEM-1 (PDB: 1NYM), NDM-1 (PDB: 4EXS), AmpC (PDB: 1C3B) y OXA-48 (PDB: 7KHQ) mediante AMDock. Los resultados del acoplamiento revelaron fuertes afinidades de unión, incluyendo la quercetina con TEM-1 (–8.9 kcal/mol), la rutina con AmpC (–9.3 kcal/mol) y NDM-1 (–6.79 kcal/mol), y el ácido gálico con OXA-48 (–7.45 kcal/mol). El perfil de interacción con BIOVIA Discovery Studio confirmó la formación de enlaces de hidrógeno, las interacciones hidrofóbicas y la complementariedad estérica. Se determinó que existe una correlación significativa entre la concentración del compuesto y la energía de enlace para TEM-1 (P = 0.023) y AmpC (P = 0.010). Las predicciones farmacocinéticas de Swiss ADME mostraron que la quercetina y el ácido gálico cumplen la regla del cinco de Lipinski, lo que indica una buena biodisponibilidad oral, a diferencia de la rutina. El análisis de huevo cocido predijo la permeabilidad de la barrera hematoencefálica para la quercetina y el ácido gálico. Las predicciones de toxicidad con ProTox-II revelaron posibles toxicidades órgano-específicas entre los ligandos principales.
En general, estos hallazgos resaltan el potencial de los fenólicos derivados de S. sediforme, particularmente la quercetina y el ácido gálico, como prometedores inhibidores multiobjetivos de β-lactamasa para combatir la resistencia a los antibióticos.
Downloads
References
1. Ghaouas, S. ; Chala, S. Cahiers Santé Médecine Thérapeutique, 2024, 33, 275-278. DOI: https://doi.org/10.1684/sanmt.2024.286
2. Maïworé, J.; Baane, M. P. ; Youssouf, D. ; Bole, J. ; Nympeye Nango, F. Y. E.S.J. 2021, 17, 107. DOI: https://doi.org/10.19044/esj.2021.v17n10p107
3. Nascimento, G. G. F.; Locatelli, J.; Freitas, P. C.; Silva, G. L. Braz. J. Microbiol. 2000, 31. DOI: https://doi.org/10.1590/S1517-83822000000400003
4. Chan, S. S.; Khoo, K. S.; Chew, K. W.; Ling, T. C.; Show, P. L. Bioresour. Technol. 2022, 344, 126159. DOI: https://doi.org/10.1016/j.biortech.2021.126159
5. Majumdar, G.; Mandal, S. Chemical Physics Impact. 2024, 8, 100501. DOI: https://doi.org/10.1016/j.chphi.2024.100501
6. Dekir, A.; Berredjem, M.; Rachedi, K. O.; Bahadi, R.; Djouad, S. E.; Bouacida, S.; Grib, I.; Iqbal, N.; Redjemia, R.; Boussaker, M. J. Mol. Struct. 2023, 1289, 135831. DOI: https://doi.org/10.1016/j.molstruc.2023.135831
7. Markham, K. R., in: Techniques of flavonoid identification (Chapters 1 & 2). Academic Press, London, 1982.
8. Bahorun, T.; Gressier, B.; Trotin, F.; Brunet, C.; Dine, T.; Luyckx, M.; Vasseur, J.; Cazin, M.; Cazin, J. C.; Pinkas, M. Arzneim.-Forsch., Beih. 1996, 46, 1086-1089.
9. Berboucha, M.; Ayouni, K.; Atmani, D.; Atmani, D.; Benboubetra, M. J. Med. Food. 2010, 13, 1-9.
10. Benyahlou, Z. D.; Baara, F. T.; Yahiaoui, S.; Megrouss, Y.; Boukabcha, N.; Djafri, A.; Chouaih, A.; Hatzidimitriou, A. J. Mol. Struct. 2023, 1277, 134781. DOI: https://doi.org/10.1016/j.molstruc.2022.134781
11. Pestana-Nobles, R.; Aranguren-Díaz, Y.; Machado-Sierra, E.; Yosa, J.; Galan-Freyle, N. J.; Sepulveda-Montaño, L. X.; Kuroda, D. G.; Pacheco-Londoño, L. C. Int. J. Mol. Sci. 2022, 23, 1630. DOI: https://doi.org/10.3390/ijms230316300
12. Lipinski, C. A. Drug Discov. 2004, 1, 337–341. DOI: https://doi.org/10.1016/j.ddtec.2004.11.007.
13. Daina, A.; Michielin, O.; Zoete, V. Sci. Rep. 2017, 7, 42717. DOI: https://doi.org/10.1038/srep42717
14. Djeridane, A.; Yousfi, M.; Nadjemi, B.; Boutassouna, D.; Stocker, P.; Vidal, N. Food Chem. 2006, 97, 654–660. DOI: https://doi.org/10.1016/j.foodchem.2005.04.028
15. Chang, C. C.; Yang, M. H.; Wen, H. M.; Chern, J. C. J. Food Drug Anal. 2020, 10. DOI: https://doi.org/10.38212/2224-6614.2748
16. Ertaş, A.; Boğa, M.; Yılmaz, M. A.; Yeşil, Y.; Haşimi, N.; Kaya, M. Ş.; Temel, H.; Kolak, U. J. Agric. Food Chem. 2014, 62, 4601‑4609. DOI: https://doi.org/10.1021/jf500067q
17. Winekenstädde, D.; Angelis, A.; Waltenberger, B.; Schwaiger, S.; Tchoumtchoua, J.; König, S.; Werz, O.; Aligiannis, N.; Skaltsounis, A. L.; Stuppner, H. Nat. Prod. Commun. 2015, 10, 83‑88.
18. Anago, E.; Winsou, I.; Moussé, W.; Fiogbé, S. Int. J. Biochem. Res. Rev. 2025, 34, 1-10.
19. Arora, S.; Nandi, S. Asian J. Pharm. Clin. Res. 2017, 10, 204–208.
20. Kitchen, D. B.; Decornez, H.; Furr, J. R.; Bajorath, J.Nat. Rev. Drug Discov. 2004, 3, 935–949. DOI: https://doi.org/10.1038/nrd1549
21. Bentz, A. B. J. Young Investig. 2009, 19.
22. Sariyer, E.; Saral, A. Turk. J. Chem. 2021, 45, 1045-1056. DOI: https://doi.org/10.3906/kim 2011 52
23. Fatima, A.; Javid, A.; Ahmed, M. JMMG. 2024, 5, 21–35. DOI: https://doi.org/10.52700/jmmg.v5i1.153
24. Bibi, Z.; Asghar, I.; Ashraf, N. M.; Zeb, I.; Rashid, U.; Hamid, A.; Ali, M. K.; Hatamleh, A. A.; Al-Dosary, M. A.; Ahmad, R.; Ali, M. Pharm. 2023, 16, Article 10. DOI: https://doi.org/10.3390/ph16101404
25. Aati, H.; Lithy, N. M.; Aati, S. Y.; Khanfar, M. A.; Hassan, H. M.; Bahr, H. S. Proc. 2025, 13, 1294. DOI: https://doi.org/10.3390/pr13051294
26. Zhang, S.; Liao, X.; Ding, T.; Ahn, J. Antibiot. 2024, 13, 260. DOI: https://doi.org/10.3390/antibiotics13030260
27. Nwokebu, G. C.; Adesina, A. B.; Isibor, C. N.; Aigbepue, S. A.; Egbo, C. C.; Pureaziba, N.; Isaac, O. O.; Owolade, A. J. J.; Alabere, H. O.; Iwuagwu, M. O.; Hussein, M. O.; Ibrahim, A.; Balogun, T. A. Bioorg. Chem. 2024, 145, 107238. DOI: https://doi.org/10.1016/j.bioorg.2024.107238
28. Kongkham, B.; Duraivadivel, P.; Hariprasad, P. J. Ethnopharmacol. 2023, 331, 118323. DOI: https://doi.org/10.1016/j.jep.2024.118323
29. Abdollahian, A.; Hazhirkamal, M.; Taheri, T.; Fatemeh, N.; Taherkhani, A. Lett. Drug. Des. Discov. 2023,20, 1086 – 1097. DOI:10.2174/1570180819666220610112231
30. Li, S.; He, H.; Parthiban, L. J.; Yin, H.; Serajuddin, A. T. M. J. Pharm. Sci. 2005, 94, 1396-1417. DOI: https://doi.org/10.1002/jps.20378
31. Strazielle, N.; Ghersi Egea, J. F. Rev. Med. Virol. 2005, 15, 105-133. https://doi.org/10.1002/rmv.454
32. Daina, A.; Zoete, V. Chem. Med. Chem. 2016, 11, 1117-1121. DOI: https://doi.org/10.1002/cmdc.201600182

Downloads
Published
Issue
Section
License
Copyright (c) 2025 Boussoualim Naouel, Krache Imane, Trabsa Hayat, Kara Anfal, Louamen Choukrane, Manallah Rahma, Benguerba Yacine

This work is licensed under a Creative Commons Attribution-NonCommercial 4.0 International License.
Authors who publish with this journal agree to the following terms:
- Authors retain copyright and grant the journal right of first publication with the work simultaneously licensed under a Creative Commons Attribution License that allows others to share the work with an acknowledgement of the work's authorship and initial publication in this journal.
- Authors are able to enter into separate, additional contractual arrangements for the non-exclusive distribution of the journal's published version of the work (e.g., post it to an institutional repository or publish it in a book), with an acknowledgement of its initial publication in this journal.