In Vitro and In Silico Studies of Bis-furyl-pyrrolo[3,4-b]pyridin-5-ones on Dengue Virus
DOI:
https://doi.org/10.29356/jmcs.v68i1.2103Keywords:
Dengue virus replicon, In vitro assays, In silico simulations, Docking, Pyrrolo[3,4-b]pyridin-5-ones, Ugi-Zhu reaction, dengue virus serotype 4Abstract
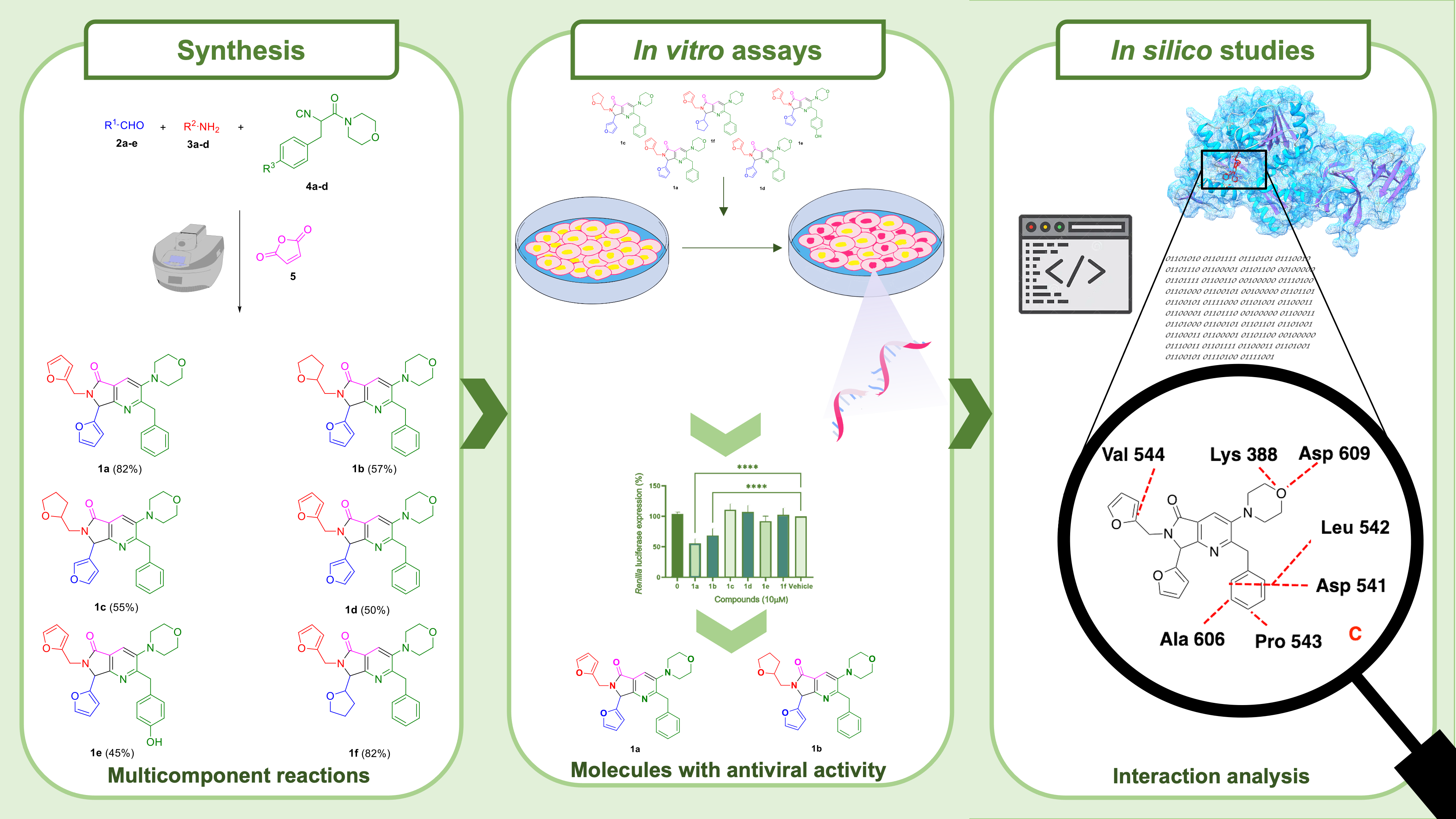
A series of six bis-furyl-pyrrolo[3,4-b]pyridin-5-ones synthesized via an Ugi-Zhu reaction coupled to a cascade process [aza Diels-Alder cycloaddition/N-acylation/aromatization] were evaluated in vitro against Dengue virus serotype 4 infection, and the Dengue virus replicon system encoding a Renilla luciferase gen reporter. Also, in silico studies on the non-structural protein 3 (NS3), a flavivirus protease comprising an attractive target for development of therapeutic antivirals bound to non-structural protein 2B (NS3-NS2B) were performed. The in vitro results showed that compounds 1a and 1b reduced the expression of Renilla luciferase in 44.2 and 31.6%, respectively. Additionally, the same compounds decreased viral load, thus revealing their potential activity against Dengue virus serotype 4. From in silico simulations, it was developed a NS3-NS2B model, which was used as a target for the studied molecules. Computational results agree with experimental data, showing that 1a is the best ligand. Finally, a pharmacophoric model was computed for NS3-NS2B, which shows that the ligands need two hydrophobic and one hydrophilic fragment. Such results suggest that two out of the six synthesized bis-furyl-pyrrolo[3,4-b]pyridin-5-ones derivatives presents potential antiviral activity against Dengue virus in vitro.
Resumen. Una serie de seis bis-furil-pirrolo[3,4-b]piridin-5-onas sintetizadas vía una reacción Ugi-Zhu acoplada a un proceso en cascada [cicloadición aza Diels-Alder/N-acilación/aromatización] fueron evaluadas in vitro contra infección por el serotipo 4 del virus del dengue y el sistema de replicón del virus del Dengue que codifica un gen reportero de la luciferasa de la Renilla. Además, se realizaron estudios in silico sobre la proteína no estructural 3 (NS3), una proteasa de flavivirus que comprende un blanco atractivo para el desarrollo de antivirales terapéuticos unidos a la proteína no estructural 2B (NS3-NS2B). Los estudios in vitro revelaron que los compuestos 1a y 1b reducen la expresión de Renilla luciferasa en un 44.2 y 31.6%, respectivamente. Adicionalmente, estos compuestos redujeron la carga viral, revelando así su actividad potencial contra el virus del Dengue serotipo 4. Derivado de las simulaciones in silico, se obtuvo un modelo homólogo para NS3-NS2B, el cual fue considerado como blanco de las moléculas estudiadas. Los resultados computacionales correlacionan con los experimentales, mostrando que 1a es el mejor ligando. Finalmente, se generó un modelo farmacofórico para NS3-NS2B, el cual muestra que los ligandos necesitan dos fragmentos hidrofóbicos y uno hidrofílico. Estos resultados demuestran que dos de los seis compuestos que se estudiaron presentan actividad antiviral in vitro.
Downloads
References
Alcaraz-Estrada, S. L.; Manzano, M. I. M.; Del Angel, R. M.; Levis, R.; Padmanabhan, R. J. Gen. Virol. 2010, 91, 2713–2718. DOI: https://doi.org/10.1099/vir.0.024083-0.
Yung, C. F.; Lee, K. S.; Thein, T. L.; Tan, L. K.; Gan, V. C.; Wong, J. G. X.; Lye, D. C.; Ng, L. C.; Leo, Y. S. Am. J. Trop. Med. Hyg. 2015, 92, 999–1005. DOI: https://doi.org/10.4269/ajtmh.14-0628.
Bhatt, S.; Gething, P. W.; Brady, O. J.; Messina, J. P.; Farlow, A. W.; Moyes, C. L.; Drake, J. M.; Brownstein, J. S.; Hoen, A. G.; Sankoh, O.; Myers, M. F.; George, D. B.; Jaenisch, T.; William Wint, G. R.; Simmons, C. P.; Scott, T. W.; Farrar, J. J.; Hay, S. I. Nature. 2013, 496, 504–507. DOI: https://doi.org/10.1038/nature12060.
Kok, B. H.; Lim, H. T.; Lim, C. P.; Lai, N. S.; Leow, C. Y.; Leow, C. H. Virus Res. 2022, 324, 199018. DOI: https://doi.org/10.1016/j.virusres.2022.199018.
Alcaraz-Estrada, S. L.; del Angel, R.; Padmanabhan, R., in: Dengue. Methods in Molecular Biology, Vol. 1138, Padmanabhan, R., Vasudevan, S., Eds., Humana Press, New York, NY, 2014, 131–150. DOI: https://doi.org/10.1007/978-1-4939-0348-1. DOI: https://doi.org/10.1007/978-1-4939-0348-1_9
Alcaraz-Estrada, S. L.; Yocupicio-Monroy, M.; Del Angel, R. M. Future Virol. 2010, 5, 575–592. DOI: https://doi.org/10.2217/FVL.10.49.
World Health Organization, https://www.who.int, accessed in June 2023.
Li, Z., Zhang, J.; Li, H. in Viral proteases, and their inhibitors, Gupta, S. P., Ed., Academic Press, 2017,163–188. DOI: https://doi.org/10.1016/B978-0-12-809712-0.00007-1.
Hannemann, H. Drug Discov. Today 2020, 25, 1026-1033. DOI: 10.1016/j.drudis.2020.03.010. DOI: https://doi.org/10.1016/j.drudis.2020.03.010
Fernandes, R. S.; Freire, M. C. L. C.; Bueno, R. V.; Godoy, A. S.; Gil, L. H. V. G.; Oliva, G. Viruses 2020, 12, 598. DOI: https://doi.org/10.3390/v12060598.
Patil, V. M.; Balasubramanian, K.; Masand, N., in: Dengue Virus Polymerase: A Crucial Target for Antiviral Drug Discovery, Gupta, S. P., Ed., Academic Press, 2019, 387–428. DOI: https://doi.org/10.1016/B978-0-12-815422-9.00014-0.
Saito, K.; Shimasaki, K.; Fukasawa, M.; Suzuki, R.; Okemoto-Nakamura, Y.; Katoh, K.; Takasaki, T.; Hanada, K. Virus Res. 2022, 322, 198935. DOI: https://doi.org/10.1016/j.virusres.2022.198935.
Jones, C. T.; Patkar, C. G.; Kuhn, R. J. Virology. 2005, 331, 247–259. DOI: https://doi.org/10.1016/j.virol.2004.10.034.
Satoh, S.; Mori, K.; Ueda, Y.; Sejima, H.; Dansako, H.; Ikeda, M.; Kato, N. Plos one. 2015, 10, e0118313. DOI: https://doi.org/10.1371/journal.pone.0118313.
Puig-Basagoiti, F.; Deas, T. S.; Ren, P.; Tilgner, M.; Ferguson, D. M.; Shi, P. Y. Antimicrob. Agents Chemother. 2005, 49, 4980–4988. DOI: https://doi.org/10.1128/aac.49.12.4980-4988.2005.
Alcaraz-Estrada, S. L.; Reichert, E. D.; Padmanabhan, R., In: Antiviral Methods and Protocols. Methods in Molecular Biology, Vol. 1030, Gong, E. Eds., Humana Press, Totowa, NJ, 2013, 283–299. DOI: https://doi.org/10.1007/978-1-62703-484-5_22.
Kato, F.; Hishiki, T. Viruses. 2016, 8, 122. DOI: 10.3390/v8050122. DOI: https://doi.org/10.3390/v8050122
Li, J. Q.; Deng, C. L.; Gu, D.; Li, X.; Shi, L.; Zhang, Q. Y.; Zhang, B.; Ye, H. Q. Antivir. Res. 2018, 150, 148–154. DOI: https://doi.org/10.1016/j.antiviral.2017.12.017.
Morales-Salazar, I.; Montes-Enríquez, F. P.; Garduño-Albino, C. E.; García-Sánchez, M. A.; Ibarra, I. A.; Rojas-Aguirre, Y.; García-Hernández, M. E.; Sarmiento-Silva, R. E.; Alcaraz-Estrada, S. L.; Díaz-Cervantes, E.; González-Zamora, E.; Islas-Jácome, A. RSC Med. Chem. 2023, 14, 154–165. DOI: https://doi.org/10.1039/d2md00350c.
Rothan, H. A.; Abdulrahman, A. Y.; Khazali, A. S.; Nor Rashid, N.; Chong, T. T.; Yusof, R. J. Pep. Sci. 2019, 25, e3196. DOI: https://doi.org/10.1002/psc.3196.
García-Ruíz, D.; Martínez-Guzmán, M. A.; Cárdenas-Vargas, A.; Marino-Marmolejo, E.; Gutiérrez-Ortega, A.; González-Díaz, E.; Morfin‑Otero, R.; Rodríguez‑Noriega, E.; Pérez‑Gómez, H.; Elizondo-Quiroga, D. Springerplus. 2016, 5, 1–9. DOI: https://doi.org/10.1186/s40064-016-2318-y.
Rao, X.; Huang, X.; Zhou, Z.; Lin, X. Biostat. Bioinforma. Biomath. 2013, 3, 71–85.
Waterhouse, A.; Bertoni, M.; Bienert, S.; Studer, G.; Tauriello, G.; Gumienny, R.; Heer, F. T.; de Beer, T. A. P.; Rempfer, C.; Bordoli, L.; Lepore, R.; Schwede, T. Nucleic Acids Res. 2018, 46, W296-W303. DOI: https://doi.org/10.1093/nar/gky427.
Hanwell, M. D.; Curtis, D. E.; Lonie, D. C.; Vandermeersch, T.; Zurek, E.; Hutchison, G. R. J. Cheminform. 2012, 4, 17. DOI: https://doi.org/10.1186/1758-2946-4-17.
Perdew, J. P.; Burke, K.; Ernzerhof, M. Phys. Rev. Lett. 1997, 78, 1396. DOI: https://doi.org/10.1103/PhysRevLett.77.3865. DOI: https://doi.org/10.1103/PhysRevLett.78.1396
Gaussian 09, Frisch, M. J.; Trucks, G. W.; Schlegel, H. B.; Scuseria, G. E.; Robb, M. A.; Cheeseman, J. R.; Scalmani, G.; Barone, V.; Mennucci, B.; Petersson, G. A.; Nakatsuji, H.; Caricato, M.; Li, X.; Hratchian, H. P.; Izmaylov, A. F.; Bloino, J.; Zheng, G.; Sonnenberg, J. L.; Hada, M.; Ehara, M.; Toyota, K.; Fukuda, R.; Hasegawa, J.; Ishida, M.; Nakajima, T.; Honda, Y.; Kitao, O.; Nakai, H.; Vreven, T.; Montgomery, J. A., Jr.; Peralta, J. E.; Ogliaro, F.; Bearpark, M.; Heyd, J. J.; Brothers, E.; Kudin, K. N.; Staroverov, V. N.; Kobayashi, R.; Normand, J.; Raghavachari, K.; Rendell, A.; Burant, J. C.; Iyengar, S. S.; Tomasi, J.; Cossi, M.; Rega, N.; Millam, J. M.; Klene, M.; Knox, J. E.; Cross, J. B.; Bakken, V.; Adamo, C.; Jaramillo, J.; Gomperts, R.; Stratmann, R. E.; Yazyev, O.; Austin, A. J.; Cammi, R.; Pomelli, C.; Ochterski, J. W.; Martin, R. L.; Morokuma, K.; Zakrzewski, V. G.; Voth, G. A.; Salvador, P.; Dannenberg, J. J.; Dapprich, S.; Daniels, A. D.; Farkas, Ö.; Foresman, J. B.; Ortiz, J. V.; Cioslowski, J.; Fox, D. J. Gaussian, Inc., Wallingford CT, 2009.
Thomsen, R.; Christensen, M. H. J. Med. Chem. 2006, 49, 3315–3321. DOI: https://doi.org/10.1021/jm051197e.
Yang, J. M.; Chen, C. C. Proteins 2004, 55, 288–304. DOI: https://doi.org/10.1002/prot.20035.
Koes, D. R.; Camacho, C. J. Nucleic Acids Res. 2012, 40, W409–W414. DOI: https://doi.org/10.1093/nar/gks378.
McAuley, A. J.; Beasley, D. W. C. in: West Nile Virus. Methods in Molecular Biology, Vol. 1435, Colpitts, T., Ed., Humana Press, New York, 2016, 19-27. DOI: https://doi.org/10.1007/978-1-4939-3670-0_3.
De Castro Barbosa, E.; Alves, T. M. A.; Kohlhoff, M.; Jangola, S. T. G.; Pires, D. E. V.; Figueiredo, A. C. C.; Alves, É. A. R.; Calzavara-Silva, C. E.; Sobral, M.; Kroon, E. G.; Rosa, L. H.; Zani C. L.; de Oliveira, J. G. Virol. J. 2022, 19, 31. DOI: https://doi.org/10.1186/s12985-022-01751-z.
Harcourt, J.; Tamin, A.; Lu, X.; Kamili, S.; Sakthivel, S. K.; Murray, J.; Queen, K.; Tao, Y.; Paden, C. R.; Zhang, J.; Li, Y.; Uehara, A.; Wang, H.; Goldsmith, C.; Bullock, H. A.; Wang, L.; Whitaker, B.; Lynch, B.; Gautam, R.; Schindewolf, C.; Lokugamage, K. G.; Scharton, D.; Plante, J. A.; Mirchandani, D.; Widen, S. G.; Narayanan, K.; Makino, S.; Ksiazek, T. G.; Plante, K. S.; Weaver, S. C.; Lindstrom, S.; Tong, S.; Menachery, V. D.; Thornburg, N. J. Emerg. Infect. Dis. 2020, 26, 1266. DOI: https://doi.org/10.3201/eid2606.200516.
Konishi, K.; Yamaji, T.; Sakuma, C.; Kasai, F.; Endo, T.; Kohara, A.; Hanada, K.; Osada, N. Front. Genet. 2022, 13, 801382. DOI: https://doi.org/10.3389/fgene.2022.801382.
Ayub, A.; Ashfaq, U. A.; Idrees, S.; Haque, A. BioResearch Open Access. 2013, 2, 392–396. DOI: https://doi.org/10.1089/biores.2013.0022
A) Lee, J.; Worrall, L. J.; Vuckovic, M.; Rosell, F. I.; Gentile, F.; Ton, A. T.; Caveney, N. A.; Ban, F.; Cherkasov, A.; Paetzel, M.; Strynadka, N. C. Nat. Commun. 2020, 11, 5877. DOI: https://doi.org/10.1038/s41467-020-19662-4. B) Lee, W. H. K.; Liu, W.; Fan, J. S.; Yang, D. Biophys. J. 2021, 120, 2444–2453. DOI: https://doi.org/10.1016/j.bpj.2021.04.015.
Soto-Acosta, R.; Jung, E.; Qiu, L.; Wilson, D. J.; Geraghty, R. J.; Chen, L. Molecules. 2021, 26, 3779. DOI: https://doi.org/10.3390/molecules26133779.

Downloads
Additional Files
Published
Issue
Section
License
Copyright (c) 2023 Ivette Morales-Salazar, Carlos E. Garduño-Albino, Flora P. Montes-Enríquez, Atilano Gutiérrez-Carrillo, Yareli Rojas-Aguirre, Nancy Viridiana Estrada-Toledo, Jorge Sandoval-Basilio, Sofía Lizeth Alcaraz-Estrada, Erik Díaz-Cervantes, Eduardo González-Zamora, Alejandro Islas-Jácome

This work is licensed under a Creative Commons Attribution-NonCommercial 4.0 International License.
Authors who publish with this journal agree to the following terms:
- Authors retain copyright and grant the journal right of first publication with the work simultaneously licensed under a Creative Commons Attribution License that allows others to share the work with an acknowledgement of the work's authorship and initial publication in this journal.
- Authors are able to enter into separate, additional contractual arrangements for the non-exclusive distribution of the journal's published version of the work (e.g., post it to an institutional repository or publish it in a book), with an acknowledgement of its initial publication in this journal.