Molecular Modelling, Docking and Pharmacokinetic Studies of N-Arylidenequinoline-3-Carbohydrazides Analogs as Novel β-Glucuronidase Inhibitors
DOI:
https://doi.org/10.29356/jmcs.v64i1.1025Keywords:
Molecular modeling, docking, GFA, β-glucuronidaseAbstract
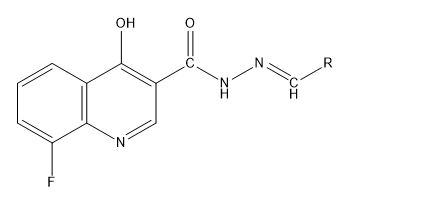
Quantitative structure-activity relationships (QSAR) modelling on 30 N-Arylidenequinoline-3-carbohydrazides analogs was performed using Multi-Linear Regression (MLR) analysis adopting Genetic Function Algorithm (GFA) method. Semi empirical method using PM6 basis set was used for complete geometry optimization of the data set. The best model was chosen based on its statistical fit due to it good internal and external validations. From the Williams plot, it can be inferred that the reported model can make prediction of new compounds that are not within the data set. The molecular docking study showed that, the most active chemical in the data set was better than the standard β-glucuronidase inhibitor both in terms of binding scores and the amino acid residues that interacted with the drug and β-glucuronidase enzyme. The Pharmacokinetic studies indicated that none of the chemicals violated any of the condition set by the Lipinski′s Rule of five which confirm the bioavailability of these chemicals. The results these findings give room for designing novel β-glucuronidase inhibitors that are highly effective.
Resumen. Se llevó a cabo la técnica de QSAR en 30 analogos de N-arilidenequinolina-3-carbohidrazidas mediante el analisis de regresesión lineal múltiple (MLS) adopatando el método del algoritmo de función genética (GFA). Para la optimización completa de la geometría del conjunto de datos se utilizó un método semiémpirico del conjunto de bases PM6. El mejor modelo fue elegido basado en función de su ajuste estadístico debido a su validación interna y externa. A partir de la gráfica de Williams, se puede inferir que el modelo reportado puede predecir nuevos compuestos que no se encuentran en el conjunto de datos. Este estudio de acomplamiento molecular mostró que, el químico más activo del conjunto de datos fue mejor que el inhibidor estándar β-glucuronidasa, tanto en términos de unión y en términos de interacción de los residuos con el fármaco y la enzima β-glucuronidasa. Los estudios farmacocinéticos que indicaron que ninguno de los fármacos incumple ninguna de las condiciones establecidas por la regla de cinco de Lipinski, en donde se confirma la biodisponibilidad de estos químicos. Los resultados de los hallazgos computacionales permiten diseñar nuevos inhibidores de la β-glucuronidasa que son altamente efectivos.
Downloads
References
Abdulfatai, U.; Uba, S.; Umar, B. A.; Ibrahim, M. T. SN Appl. Sci. 2019, 1, 499. DOI: https://doi.org/10.1007/s42452-019-0512-6
Abdullahia, M.; Shallangwaa, G. A.; Ibrahima, M. T.; Bello, A.U.; Arthura, D. E.; Uzairua, A.; Mamzaa, P. J. King Saud Univ., Sci. 2018.
Abdullahia, M.; Shallangwaa,G. A.; Ibrahima, M. T.; Bello, A. U.; Arthura, D. E.; Uzairua, A.; Mamzaa, P. JOTCS 2018 , 5, 1387-1398.
Adedirin, O.; Uzairu, A.; Shallangwa, G. A.; Abechi, S. E. jCEC 2018, 4, 0065-0084. DOI: https://doi.org/10.18540/jcecvl4iss1pp0065-0084
Ali, F.; Khan, K. M.; Salar, U.; Iqbal, S.; Taha, M.; Ismail, N. H.; Perveen, S.; Wadood, A.; Ghufran, M.; Ali, B. Bioorg. Med. Chem. 2016, 24, 3624–3635. DOI: https://doi.org/10.1016/j.bmc.2016.06.002
Ambure, P.; Aher, R. B.; Gajewicz, A.; Puzyn, T.; Roy, K. Chemom. Intell. Lab. Syst. 2015 , 147, 1-13. DOI: https://doi.org/10.1016/j.chemolab.2015.07.007
Amin, S. A.; Gayen, S. JTUSCI 2016, 10, 896-905. DOI: https://doi.org/10.1016/j.jtusci.2016.04.009
Arthur, D. E.; Uzairu, A.; Mamza, P.; Abechi, S. E.; Shallangwa, G. BJBAS 2016,5, 320-333. DOI: https://doi.org/10.1016/j.bjbas.2016.11.003
Beaud, D.; Tailliez, P.; Anba-Mondoloni, J. Microbiology 2005, 151, 2323-2330. DOI: https://doi.org/10.1099/mic.0.27712-0
Beheshti, A.; Pourbasheer, E.; Nekoei, M.; Vahdani, S. J. Saudi Chem. Soc. 2016, 20, 282-290. DOI: https://doi.org/10.1016/j.jscs.2012.07.019
Davis, A. M.; Riley, R. J. Curr. Opin. Chem. Biol. 2004, 8, 378-386. DOI: https://doi.org/10.1016/j.cbpa.2004.06.005
De Moreno De Leblana, A.; Perdigón, G. Biocell 2005, 29, 15-24.
Gloux, K.; Berteau, O.; Béguet, F.; Leclerc, M.; Doré, J. Proc. Natl. Acad. Sci. U.S.A. 2011 ,108, 4539-4546. DOI: https://doi.org/10.1073/pnas.1000066107
Ibrahim, M.T; Uzairu, A.; Shallangwa, G.A.; Ibrahim A. jCEC 2018, 4, 0276-0285. DOI: https://doi.org/10.18540/jcecvl4iss2pp0276-0285
Ibrahim, M.T.; Uzairu, A.; Shallangwaa, G. A. J. King Saud Univ. Sci. 2018.
Ismail, S.Y.; Uzairu, A; Sagagi, B.; Sabiu, M. JOTCSA 2018, 5, 1337-1350. DOI: https://doi.org/10.18596/jotcsa.449778
Jalali-Heravi, M.; Kyani, A. J. Chem. Inf Model. 2004, 44, 1328-1335. DOI: https://doi.org/10.1021/ci0342270
Jorgensen, W.L. AAAS 2004, 303, 1813-1818. DOI: https://doi.org/10.1126/science.1096361
Kennard, R.W.; Stone, L. A. Technometrics 1969, 11, 137-148. DOI: https://doi.org/10.1080/00401706.1969.10490666
Khan, K. M.; Taha, M.; Rahim, F.; Fakhri, M. I.; Jamil, W.; Khan, M.; Rasheed, S.; Karim, A.; Perveen, S.; Iqbal, M. J. Chem. Soc. Pak. 2013, 34.
Kitchen, D.B.; Decornez, H.; Furr, J. R.; Bajorath, J. Nat. Rev. Drug Discovery 2004, 3, 935. DOI: https://doi.org/10.1038/nrd1549
Lipinski, C. A.; Lombardo, F.; Dominy, B. W.; Feeney, P.J. Adv. Drug Delivery Rev. 1997, 23, 3-25. DOI: https://doi.org/10.1016/S0169-409X(96)00423-1
Ojha Lokendra, K.; Rachana, S.; Rani, B. M. Int J. Res. Biosci Plan Biol. 2013, 2, 1-12.
Salar, U.; Taha, M.; Ismail, N.H; Khan, K. M.; Imran,S.; Perveen, S.; Wadood, A.; Riaz, M. Bioorg. Med. Chem. 2016, 24, 1909-1918. DOI: https://doi.org/10.1016/j.bmc.2016.03.020
Sharma, R.; Patil, S.; Maurya, P. SAR QSAR Environ. Res. 2014, 25, 189-203. DOI: https://doi.org/10.1080/1062936X.2013.875484
Taha, M.; Ismail, N. H.; Imran, S.; Selvaraj, M.; Rashwan, H.; Farhanah, F. U.; Rahim, F.; Kesavanarayanan, K. S.; M. Ali, M. Bioorg. Chem. 2015, 61, 36-44. DOI: https://doi.org/10.1016/j.bioorg.2015.05.010
M. Taha, M.; Sultan, S.; Nuzar, H. A.; Rahim, F.; Imran, S.; Ismail, N.H.; Naz, H.; Ullah, H. Bioorg. Med. Chem. 2016, 24, 3696-3704. DOI: https://doi.org/10.1016/j.bmc.2016.06.008
Taha, M.; Ullah, H.; Al Muqarrabun, L. M. R.; Khan, M. N.; Rahim, F.; Ahmat, N.; Ali, M.; Perveen, S. Eur. J. Med. Chem. 2018, 143, 1757-1767. DOI: https://doi.org/10.1016/j.ejmech.2017.10.071
Tropsha, A.; Bajorath, J. R. JACS 2016, 59. DOI: https://doi.org/10.1021/acs.jmedchem.5b01945
Tropsha, A.; Gramatica, P.; Gombar, V. K. Mol. Inf. 2003, 22, 69-77. DOI: https://doi.org/10.1002/qsar.200390007
Veerasamy, R.; Rajak, H.; Jain, A.; Sivadasan, S.; Varghese, C.P.; Agrawal, R. K. Int. J. Drug Des. Discovery, 2011, 3, 511-519.
Yap, C.W. J. Comput. Chem. 2011, 32, 1466-1474. DOI: https://doi.org/10.1002/jcc.21707

Downloads
Additional Files
Published
Issue
Section
License
Authors who publish with this journal agree to the following terms:
- Authors retain copyright and grant the journal right of first publication with the work simultaneously licensed under a Creative Commons Attribution License that allows others to share the work with an acknowledgement of the work's authorship and initial publication in this journal.
- Authors are able to enter into separate, additional contractual arrangements for the non-exclusive distribution of the journal's published version of the work (e.g., post it to an institutional repository or publish it in a book), with an acknowledgement of its initial publication in this journal.