TEN ELEVEN TRANSLOCATION 2 (TET2) Improves the Adipogenic Potential of Dental Pulp Stem Cells
DOI:
https://doi.org/10.29356/jmcs.v67i3.2057Palabras clave:
Adipogenesis, cellular differentiation, dental pulp stem cells, DNA demethylation, TET2Resumen
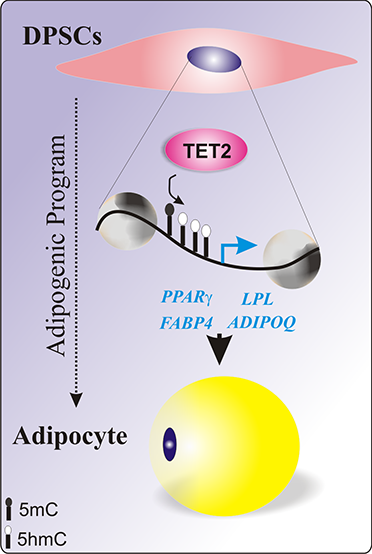
Abstract. Although mesenchymal stem cells (MSCs) are the major source of adipocytes, adipogenesis is a highly complex process whose mechanisms driving adipocyte origin and development remain poorly understood. Previous findings by our group have shown that different MSCs from the oral cavity displayed differential expression of TET2, a key regulator of DNA methylation, during adipogenic induction. Therefore, we proposed to evaluate the effects of the overexpression of TET2 on the adipogenic response of a cell line with a low natural commitment to this cell fate. We used human dental pulp cells, which were characterized through flow cytometry for mesenchymal markers, analysis of stemness-related genes (OCT4, NANOG, SOX2, KLF4 and c-MYC) and trilineage capacity. The characterized cells were transfected with TET2 and induced to adipogenesis for 21 days. Our results show that TET2-overexpressing cells (pTET2-OE cells) exhibit an earlier adipogenic response. In addition, pTET2-OE cells induced more than 4-, 2.5-, 30-, and 50-fold expression of the adipogenic markers PPARg, ADIPOQ, FABP4, and LPL, respectively. Our findings suggest that TET2 overexpression could induce demethylation of the PPARg locus, the master regulator of adipogenesis, and of the other adipogenic genes, improving the transition of dental pulp stem cells toward adipogenic commitment.
Resumen. Aunque las células troncales mesenquimales (MSC) son la principal fuente de adipocitos, la adipogénesis es un proceso complejo cuyos mecanismos que impulsan el origen y desarrollo de los adipocitos permanecen sin conocerse completamente. Previamente nuestro grupo ha demostrado que diferentes MSC de origen bucal mostraron una expresión diferencial de TET2, un regulador clave de la metilación del ADN, durante la inducción adipogénica. Por lo tanto, se propuso evaluar el efecto de la sobreexpresión de TET2 en la respuesta adipogénica en una línea celular con bajo compromiso hacia la diferenciación adipogénica. Nosotros usamos células de la pulpa dental las cuales fueron caracterizadas mediante citometría de flujo para marcadores mesenquimales, análisis de genes de pluripotencia (OCT4, NANOG, SOX2, KLF4 and c-MYC) y capacidad tri-linaje. Las células caracterizadas fueron transfectadas con TET2 e inducidas a la adipogénesis por 21 días. Nuestros hallazgos demuestran que las células que sobre expresan TET2 (pTET-OE) muestran una respuesta adipogénica más temprana. Además, las células pTET-OE incrementaron más de 4-, 2.5-, 30-, y 50 veces la expresión de los marcadores adipogénicos PPARg, ADIPOQ, FABP4 y LPL respectivamente. Nuestros resultados sugieren que la sobreexpresión de TET2 podría inducir la desmetilación del locus de PPARg, el regulador maestro de la adipogénesis y de los genes adipogénicos, lo que mejora la transición de las células troncales de la pulpa dental hacia el compromiso adipogénico.
Descargas
Citas
Shi, X.; Mao, J.; Liu, Y. Stem. Cells Transl. Med. 2020, 9, 445-464. DOI:10.1002/sctm.19-0398. DOI: https://doi.org/10.1002/sctm.19-0398
Karaoz, E.; Dogan, B. N.; Aksoy, A.; Gacar, G.; Akyuz, S.; Ayhan, S.; Genc, Z.S.; Yuruker, S.; Duruksu, G.; Demircan, P.C.; et al. Histochem. Cell. Biol. 2010, 133, 95-112. DOI:10.1007/s00418-009-0646-5. DOI: https://doi.org/10.1007/s00418-009-0646-5
De la Rosa-Ruiz, M. D. P.; Alvarez-Perez, M. A.; Cortes-Morales, V. A.; Monroy-Garcia, A.; Mayani, H.; Fragoso-Gonzalez, G.; Caballero-Chacon, S.; Diaz, D.; Candanedo-Gonzalez, F.; Montesinos, J. J. Cells. 2019, 8. DOI:10.3390/cells8121491. DOI: https://doi.org/10.3390/cells8121491
Kobayashi, T.; Torii, D.; Iwata, T.; Izumi, Y.; Nasu, M.; Tsutsui, T. W. Hum. Cell. 2020, 33, 490-501. DOI:10.1007/s13577-020-00327-9. DOI: https://doi.org/10.1007/s13577-020-00327-9
Kang, R.; Zhou, Y.; Tan, S.; Zhou, G.; Aagaard, L.; Xie, L.; Bunger, C.; Bolund, L.; Luo, Y. Stem. Cell. Res. Ther. 2015, 6, 144. DOI:10.1186/s13287-015-0137-7. DOI: https://doi.org/10.1186/s13287-015-0137-7
Rauch, A.; Haakonsson, A. K.; Madsen, J. G. S.; Larsen, M.; Forss, I.; Madsen, M. R.; Van Hauwaert, E. L.; Wiwie, C.; Jespersen, N. Z.; Tencerova, M.; et al. Nat. Genet. 2019, 51, 716-727. DOI: 10.1038/s41588-019-0359-1. DOI: https://doi.org/10.1038/s41588-019-0359-1
Ponnaiyan, D.; Jegadeesan, V. Eur. J. Dent. 2014, 8, 307-313. DOI:10.4103/1305-7456.137631. DOI: https://doi.org/10.4103/1305-7456.137631
Mercado-Rubio, M. D.; Perez-Argueta, E.; Zepeda-Pedreguera, A.; Aguilar-Ayala, F. J.; Penaloza-Cuevas, R.; Ku-Gonzalez, A.; Rojas-Herrera, R. A.; Rodas-Junco, B. A.; Nic-Can, G. I. J. Pers. Med. 2021, 11. DOI:10.3390/jpm11080738. DOI: https://doi.org/10.3390/jpm11080738
Fajas, L.; Egler, V.; Reiter, R.; Hansen, J.; Kristiansen, K.; Debril, M.-B.; Miard, S.; Auwerx, J. Developmental Cell. 2002, 3, 903-910. DOI:10.1016/s1534-5807(02)00360-x. DOI: https://doi.org/10.1016/S1534-5807(02)00360-X
Fracaro, L.; Senegaglia, A. C.; Herai, R. H.; Leitolis, A.; Boldrini-Leite, L. M.; Rebelatto, C. L. K.; Travers, P. J.; Brofman, P. R. S.; Correa, A. Int. J. Mol. Sci. 2020, 21. DOI:10.3390/ijms21082753. DOI: https://doi.org/10.3390/ijms21082753
Argaez-Sosa, A. A.; Rodas-Junco, B. A.; Carrillo-Cocom, L. M.; Rojas-Herrera, R. A.; Coral-Sosa, A.; Aguilar-Ayala, F. J.; Aguilar-Perez, D.; Nic-Can, G. I. Front. Cell Dev. Biol. 2022, 10, 791667. DOI:10.3389/fcell.2022.791667. DOI: https://doi.org/10.3389/fcell.2022.791667
Serandour, A. A.; Avner, S.; Oger, F.; Bizot, M.; Percevault, F.; Lucchetti-Miganeh, C.; Palierne, G.; Gheeraert, C.; Barloy-Hubler, F.; Peron, C. L.; et al. Nucleic Acids Res. 2012, 40, 8255-8265. DOI:10.1093/nar/gks595. DOI: https://doi.org/10.1093/nar/gks595
Yoo, Y.; Park, J. H.; Weigel, C.; Liesenfeld, D. B.; Weichenhan, D.; Plass, C.; Seo, D. G.; Lindroth, A. M.; Park, Y. J. Int. J. Obes. (Lond.). 2017, 41, 652-659. DOI:10.1038/ijo.2017.8. DOI: https://doi.org/10.1038/ijo.2017.8
Hou, Y.; Zhang, Z.; Wang, Y.; Gao, T.; Liu, X.; Tang, T.; Du, Q. Gene. 2020, 733, 144265. DOI: 10.1016/j.gene.2019.144265. DOI: https://doi.org/10.1016/j.gene.2019.144265
Li, N.; Liu, L.; Liu, Y.; Luo, S.; Song, Y.; Fang, B. Mol. Ther. Nucleic Acids. 2020, 19, 619-626. DOI: 10.1016/j.omtn.2019.12.017. DOI: https://doi.org/10.1016/j.omtn.2019.12.017
Cakouros, D.; Hemming, S.; Gronthos, K.; Liu, R.; Zannettino, A.; Shi, S.; Gronthos, S. Epigenetics Chromatin. 2019, 12, 3. DOI:10.1186/s13072-018-0247-4. DOI: https://doi.org/10.1186/s13072-018-0247-4
Gopinathan, G.; Kolokythas, A.; Luan, X.; Diekwisch, T.G. Stem Cells Dev. 2013, 22, 1763-1778. DOI:10.1089/scd.2012.0711. DOI: https://doi.org/10.1089/scd.2012.0711
Livak, K. J.; Schmittgen, T. D. Methods. 2001, 25, 402-408. DOI:10.1006/meth.2001.1262. DOI: https://doi.org/10.1006/meth.2001.1262
Dominici, M.; Le Blanc, K.; Mueller, I.; Slaper-Cortenbach, I.; Marini, F.; Krause, D.; Deans, R.; Keating, A.; Prockop, D.; Horwitz, E. Cytotherapy. 2006, 8, 315-317. DOI:10.1080/14653240600855905. DOI: https://doi.org/10.1080/14653240600855905
Ma, L.; Makino, Y.; Yamaza, H.; Akiyama, K.; Hoshino, Y.; Song, G.; Kukita, T.; Nonaka, K.; Shi, S.; Yamaza, T. PLoS One 2012, 7, e51777. DOI: 10.1371/journal.pone.0051777. DOI: https://doi.org/10.1371/journal.pone.0051777
Padial-Molina, M.; de Buitrago, J. G.; Sainz-Urruela, R.; Abril-Garcia, D.; Anderson, P.; O'Valle, F.; Galindo-Moreno, P. Int. J. Mol. Sci. 2019, 20. DOI:10.3390/ijms20092171. DOI: https://doi.org/10.3390/ijms20092171
Paino, F.; La Noce, M.; Giuliani, A.; De Rosa, A.; Mazzoni, S.; Laino, L.; Amler, E.; Papaccio, G.; Desiderio, V.; Tirino, V. Clin. Sci. (Lond.). 2017, 131, 699-713. DOI:10.1042/CS20170047. DOI: https://doi.org/10.1042/CS20170047
Duncan, H. F.; Smith, A. J.; Fleming, G. J.; Moran, G. P.; Cooper, P. R. Genom. Data. 2015, 5, 391-393. DOI: 10.1016/j.gdata.2015.07.013. DOI: https://doi.org/10.1016/j.gdata.2015.07.013
Pekovits, K.; Kropfl, J. M.; Stelzer, I.; Payer, M.; Hutter, H.; Dohr, G. Histochem. Cell Biol. 2013, 140, 611-621. DOI: 10.1007/s00418-013-1140-7. DOI: https://doi.org/10.1007/s00418-013-1140-7
Monterubbianesi, R.; Bencun, M.; Pagella, P.; Woloszyk, A.; Orsini, G.; Mitsiadis, T. A. Sci. Rep. 2019, 9, 1761. DOI: 10.1038/s41598-018-37981-x. DOI: https://doi.org/10.1038/s41598-018-37981-x
Wu, X.; Zhang, Y. Nat. Rev. Genet. 2017, 18, 517-534. DOI:10.1038/nrg.2017.33. DOI: https://doi.org/10.1038/nrg.2017.33
Yang, Y. K.; Ogando, C. R.; Wang See, C.; Chang, T. Y.; Barabino, G. A. Stem. Cell Res. Ther. 2018, 9, 131. DOI:10.1186/s13287-018-0876-3. DOI: https://doi.org/10.1186/s13287-018-0876-3
Feng, L.; Zhou, J.; Xia, B.; Tian, B. F. Cell Reprogram. 2020, 22, 3-13. DOI:10.1089/cell.2019.0045. DOI: https://doi.org/10.1089/cell.2019.0045
Rosen, E.D.; MacDougald, O.A. Nat. Rev. Mol. Cell. Biol. 2006, 7, 885-896. DOI:10.1038/nrm2066. DOI: https://doi.org/10.1038/nrm2066
Xi, Y.; Shen, W.; Ma, L.; Zhao, M.; Zheng, J.; Bu, S.; Hino, S.; Nakao, M. Biochem. Biophys. Res. Commun. 2016, 472, 617-623. DOI: 10.1016/j.bbrc.2016.03.015. DOI: https://doi.org/10.1016/j.bbrc.2016.03.015
Gupta, R. K. Curr. Biol. 2014, 24, R988-993. DOI: 10.1016/j.cub.2014.09.003. DOI: https://doi.org/10.1016/j.cub.2014.09.003
Matsushita, K.; Dzau, V. J. Lab. Invest. 2017, 97, 1158-1166. DOI: 10.1038/labinvest.2017.42. DOI: https://doi.org/10.1038/labinvest.2017.42

Descargas
Archivos adicionales
Publicado
Número
Sección
Licencia
Derechos de autor 2023 Jose Alejandro Balam-Lara , Leydi Maribel Carrillo-Cocom, Beatriz Rodas-Junco , Liliana Villanueva-Lizama, Geovanny Nic-Can

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Authors who publish with this journal agree to the following terms:
- Authors retain copyright and grant the journal right of first publication with the work simultaneously licensed under a Creative Commons Attribution License that allows others to share the work with an acknowledgement of the work's authorship and initial publication in this journal.
- Authors are able to enter into separate, additional contractual arrangements for the non-exclusive distribution of the journal's published version of the work (e.g., post it to an institutional repository or publish it in a book), with an acknowledgement of its initial publication in this journal.